Nathaniel T. Jeanson, Ph.D.
Dr. Jeanson je vědecký pracovník, Ph.D. obdržel na Harvardově univerzitě v oblasti vývojové a buněčné biologie
(Z časopisu Acts and Facts a http://www.icr.org/i/pdf/af/af1101.pdf přeložil Pavel Kábrt – 2/2011)
Tým ICR, který se zabývá vědami o životě (Life Sciences), rozpoznal několik klíčových výzkumných cílů v oblasti biologie a vzniku organizmů. Naším současným úkolem je zpracovat literaturu týkající se evolučního stromu života (1) se zřetelem na práce týkající se molekulárních dat. (2, 3) Zvláště se věnujeme prvotním výsledkům a všímáme si nespojitostí (diskontinuit). (3)
Pro srovnání druhů na molekulární úrovni je třeba získat sekvence DNA nebo proteinů daných druhů. Tyto informace získáváme ze dvou zdrojů:
- Nových laboratorních experimentů prováděných výzkumníkem
- Veřejně dostupných dat z již publikovaných experimentů
Ten druhý způsob je velmi bohatým zdrojem pro srovnávání, protože už existuje mnoho zjištěných biologických sekvencí. Dohromady oba zdroje tedy představují hlavní materiál pro porovnávání různých druhů organizmů.
Když už máme k dispozici DNA nebo proteinové sekvence různých druhů organizmů, existuje mnoho způsobů, jak je molekulárně porovnat. Jeden z nejběžnějších způsobů je založen na genových sekvenčních rozdílech mezi organizmy. Protože různé organizmy mají často nějaký společný základní genový potenciál, a sekvence těchto společných genů se často významně odlišují (vzhledem k danému genu), jednotliví tvorové jsou rozpoznatelní právě na základě těchto unikátních genových sekvencí. Kvantifikace sekvenčních rozdílů mezi druhy je velmi mocný nástroj pro jejich srovnávání.
Existuje celá škála metod, jak rozdíly v genových sekvencích můžeme změřit napříč jednotlivými druhy. Jedna z těchto metod, kterou jsme použili, je založena na softwarovém programu CLUSTAL W, který je volně přístupný online na EBI – European Bioinformatics Institute. Tento program srovnává vložené sekvence v jejich celé délce každé z nich a hledá optimální přiřazení na základě parametrů vložených do programu. Poté, co porovná různé sekvence v jednom kroku, EBI verze programu CLUSTAL W nejprve provede srovnání po párech (přiřazení u všech sekvencí jednoho páru ke druhému v daném čase), dřív než vyhotoví sestavení všech porovnávaných sekvencí. Po optimalizaci porovnání jsou jedním z výsledků CLUSTAL W parametry procentuální identity mezi páry vložených sekvencí. Tento parametr je přímým zápisem sekvenčních rozdílů mezi jednotlivými tvory. Tím, že provedeme porovnávání párů napříč různými druhy, můžeme pak jednotlivé druhy řadit a odlišit na základě sekvenčních odlišností jeden od druhého.
Jeden z možných příkladů takových výsledků porovnání pomocí CLUSTAL W je zobrazen níže. Čísla v jednotlivých buňkách tabulky zobrazují jednotlivé hodnoty porovnání párů u druhů, které jsou jmenovány na začátku sloupců a řádků; jsou to procentuální totožnosti mezi genovými sekvencemi dvou srovnávaných druhů. V této jedné z možných tabulek vykazuje každý tvor hierarchickou procentuální identitu mezi ostatními organizmy. Tento typ výsledku je užitečný pro klasifikaci jednotlivých tvorů a vytvoření jejich vztahů k ostatnímu stvoření.
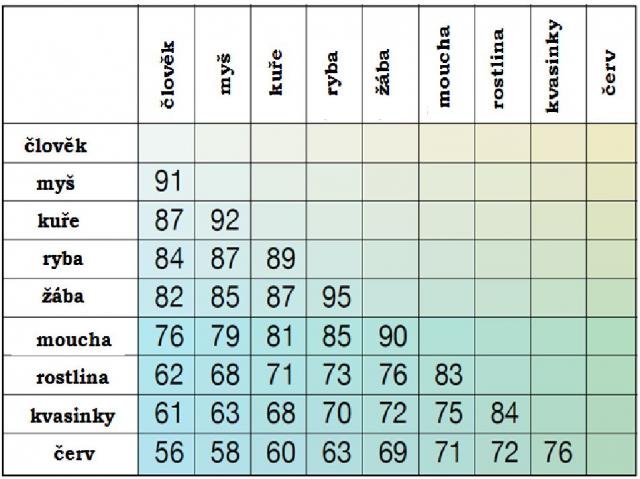
Závěrem: porovnávání molekulárních sekvencí je velmi užitečný způsob, jak analyzovat a kategorizovat rozdíly mezi různými organizmy, a očekáváme, že tato srovnávání jasně prokážou mezery v přírodě, jak to předpokládá Genesis 1.
Předchozí molekulární srovnání mezi tvory ukázaly rozdíly mezi druhy, které by Darwin nikdy nepředpokládal. Očekávejte další fascinující výsledky v následujících vydáních Acts and Facts.
Poznámka Pavla Kábrta: doporučuji při pohledu na uvedenou tabulku vybrat si nejprve nějaké blízké číselné údaje (třeba 91-90, 84-87 atd.) a najít si je všechny v tabulce a porovnat organizmy, které jsou si blízko v procentuální identitě porovnávaných sekvencí. Je to výsměch evoluční teorii!
Odkazy:
1. Jeanson, N. 2010. Literature Review: Simplifying the Research
Process. Acts & Facts. 39 (11): 6.
2. Jeanson, N. 2010. New Frontiers in
Animal Classification. Acts & Facts.
39 (5): 6.
3. Jeanson, N. 2010. Literature Review:
Molecular Data and the Tree
of Life. Acts & Facts. 39 (12): 6.

